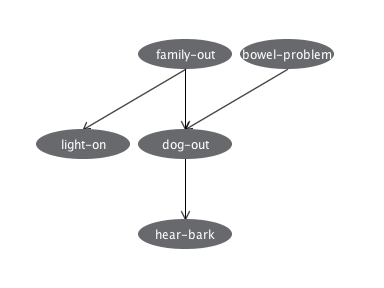
前回のFamily Out Problemの確率モデル(下図)を題材に、ナイーブベイズ分類器を使ってみる。
Family Out Problem
この問題において、family-out以外の変数の真偽値が与えられた時にfamily-out=TRUEかどうかを判定するよう、ナイーブベイズ分類器を学習させてみる。
import numpy as np
import matplotlib.pyplot as plt
from sklearn.naive_bayes import BernoulliNB
from sklearn.metrics import precision_recall_fscore_support
from sklearn.metrics import roc_curve, auc
def generate_sample():
"Sample data generator of the Family Out problem"
fo = np.random.binomial(1, 0.15)
bp = np.random.binomial(1, 0.01)
lo = np.random.binomial(1, (0.05, 0.6)[fo])
do = np.random.binomial(1, ((0.3, 0.97), (0.9, 0.99))[fo][bp])
hb = np.random.binomial(1, (0.01, 0.7)[do])
return [fo, bp, lo, do, hb]
# Generate training data and test data
train_data = np.array([generate_sample() for _ in range(1000)])
test_data = np.array([generate_sample() for _ in range(100)])
X_train = train_data[:, 1:5] # other than fo
y_train = train_data[:, 0] # only fo
X_test = test_data[:, 1:5] # other than fo
y_test = test_data[:, 0] # only fo
# Train a Naive Bayes classifier
clf = BernoulliNB()
clf.fit(X_train, y_train)
# Evaluate with training data
y_pred = clf.predict(X_train)
metrics = precision_recall_fscore_support(y_train, y_pred)
print('Evaluation with training data')
print('Class FamilyOut=False: Precision={:.3f}, Recall={:.3f}, F-measure={:.3f}'.format(metrics[0][0], metrics[1][0], metrics[2][0]))
print('Class FamilyOut=True : Precision={:.3f}, Recall={:.3f}, F-measure={:.3f}'.format(metrics[0][1], metrics[1][1], metrics[2][1]))
# Evaluate with test data
y_pred = clf.predict(X_test)
metrics = precision_recall_fscore_support(y_test, y_pred)
print('Evaluation with test data')
print('Class FamilyOut=False: Precision={:.3f}, Recall={:.3f}, F-measure={:.3f}'.format(metrics[0][0], metrics[1][0], metrics[2][0]))
print('Class FamilyOut=True : Precision={:.3f}, Recall={:.3f}, F-measure={:.3f}'.format(metrics[0][1], metrics[1][1], metrics[2][1]))
# Draw ROC curve
y_train_post = clf.predict_proba(X_train)[:, 0]
y_test_post = clf.predict_proba(X_test)[:, 0]
fpr, tpr, thresholds = roc_curve(y_train, y_train_post, pos_label=0)
roc_auc = auc(fpr, tpr)
plt.plot(fpr, tpr, 'k-', lw=2, label='ROC for training data (area = {:.2f})'.format(roc_auc))
fpr, tpr, thresholds = roc_curve(y_test, y_test_post, pos_label=0)
roc_auc = auc(fpr, tpr)
plt.plot(fpr, tpr, 'k--', lw=2, label='ROC for test data (area = {:.2f})'.format(roc_auc))
plt.xlabel('False Positive Rate')
plt.ylabel('True Positive Rate')
plt.title('ROC curve of BernoulliNB')
plt.legend(loc="lower right")
plt.show()
問題のモデルに従って学習用データとテストデータをランダムに生成し、sklearn.naive_bayes.BernoulliNBの分類器を学習させ、テストデータを分類させ、family-out=FALSEとfamily-out=TRUEのそれぞれについてPrecision, Recall, F値を計算している。
加えて、予測したfamily-out=FALSE/TRUEの確率から、ROC曲線とAUCを出力している。
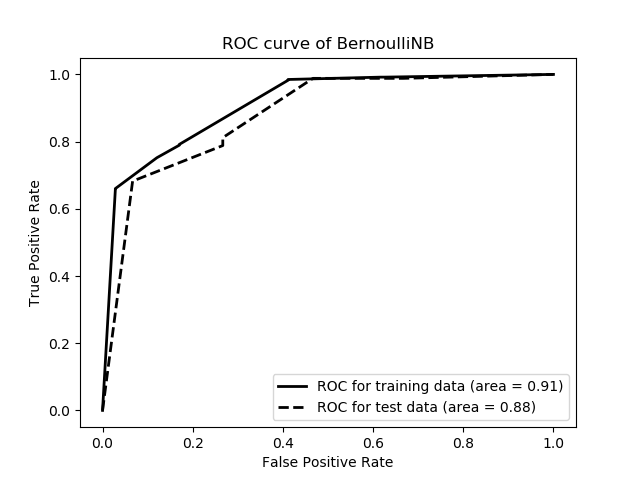
出力例
ROC曲線Evaluation with training data Class FamilyOut=False: Precision=0.936, Recall=0.983, F-measure=0.959 Class FamilyOut=True : Precision=0.847, Recall=0.589, F-measure=0.695 Evaluation with test data Class FamilyOut=False: Precision=0.923, Recall=0.988, F-measure=0.955 Class FamilyOut=True : Precision=0.889, Recall=0.533, F-measure=0.667
その時のtraining_dataとtest_data(.arff形式)
family_out_train.arff
family_out_test.arff
ナイーブベイズ分類器には、同じクラスのデータでは全ての特徴が独立に出現する、つまりbp,lo,do,hbに依存関係が無く、これらがTRUEになる確率はfoの値だけで決まるという仮定があるが、この問題ではbp,do,hbに依存関係があるので、ベイジアンネットワークの方がうまく学習できると考えられる。
なのでベイジアンネットワークで分類した場合の性能と比較したいが、scikit-learn(0.18)にベイジアンネットワークが無いので、代わりにWeka 3.8.1のBayesNetを用いて学習用データで学習させ、テストデータを分類した結果と比較してみる。
Wekaの操作手順
- Weka ExplorerのPreprocessタブでOpen file...ボタンを押し、family_out_train.arffを開く
- ClassifyタブでClassifierとしてBayesNetを選択する
- "BayesNet -D ..."とあるフィールドをクリックしてパラメーター設定画面を開き、searchAlgorithmのフィールドをクリックして、initAsNaiveBayes=False, maxNrOfParents=2に変更
- Test optionsのSupplied test setのボタンを押し、family_out_test.arffを開き、Classとしてfamily-outを選択
- クラスをfamily-outを選択し、Startボタンを押す
=== Run information ===
Scheme: weka.classifiers.bayes.BayesNet -D -Q weka.classifiers.bayes.net.search.local.K2 -- -P 2 -N -S BAYES -E weka.classifiers.bayes.net.estimate.SimpleEstimator -- -A 0.5
=== Detailed Accuracy By Class ===
TP Rate FP Rate Precision Recall F-Measure MCC ROC Area PRC Area Class
0.988 0.467 0.923 0.988 0.955 0.651 0.889 0.967 0
0.533 0.012 0.889 0.533 0.667 0.651 0.889 0.648 1
Weighted Avg. 0.920 0.398 0.918 0.920 0.911 0.651 0.889 0.919
=== Confusion Matrix ===
a b <-- classified as
84 1 | a = 0
7 8 | b = 1
BernoulliNBのテストデータに対する出力と、Precision, Recall, F-measureが完全に一致している。
WekaのBayesNetのデフォルト設定では正しいネットワークが学習されなかったが、上記手順の3.のように設定を変えると、大体次のように正しいネットワークになった。
BernoulliNBの性能には試行毎にばらつきがあったが、1000回の平均を取ってみたのが次の値であり、上記の結果は特別に良い例ではない。
また、計10回、同じデータでWekaのBayesNetの性能と比較した所、Precision, Recall, F-measureについては、8回は一致していた。Average with training data Class FamilyOut=False: Precision=0.925, Recall=0.982, F-measure=0.952 Class FamilyOut=True : Precision=0.844, Recall=0.545, F-measure=0.661 Average with test data Class FamilyOut=False: Precision=0.925, Recall=0.982, F-measure=0.952 Class FamilyOut=True : Precision=0.842, Recall=0.544, F-measure=0.651 Average of AUC with training data=0.894 Average of AUC with test data=0.895
ROC曲線のAUCはWekaのBayesNetの方が高いことが多かったが、BernoulliNBでも大体0.85-0.90の範囲であり、十分に高かった。
従って、この確率モデルに対して、ナイーブベイズ分類器は予測性能が十分に高いと言える。
なお、ここまでの結果では、学習データに対する各種性能値とテストデータに対する値にほぼ差が無いが、これは学習データのサンプル数が1000と十分に多く、偏りが無いからである。これを100にすると、次のように、テストデータに対する成績が少し下がるが、それでも、学習データだけに対して大幅に成績が良い「過学習(overfitting)」の状態と言える程ではない。
学習データ数が100の1000試行の平均
Average with training data Class FamilyOut=False: Precision=0.931, Recall=0.963, F-measure=0.945 Class FamilyOut=True : Precision=0.793, Recall=0.582, F-measure=0.650 Average with test data Class FamilyOut=False: Precision=0.926, Recall=0.955, F-measure=0.938 Class FamilyOut=True : Precision=0.767, Recall=0.564, F-measure=0.620 Average of AUC with training data=0.899 Average of AUC with test data=0.890
なお、上記のコードにおいて、
の行をclf = BernoulliNB()
に変えると、次のように、学習データに対しても全く分類されなくなってしまう。clf = MultinomialNB()
Evaluation with training data Class FamilyOut=False: Precision=0.851, Recall=1.000, F-measure=0.920 Class FamilyOut=True : Precision=0.000, Recall=0.000, F-measure=0.000 Evaluation with test data Class FamilyOut=False: Precision=0.850, Recall=1.000, F-measure=0.919 Class FamilyOut=True : Precision=0.000, Recall=0.000, F-measure=0.000
としても同様(0.01刻みで0.01〜1.0まで試した)、clf = MultinomialNB(alpha=0.5)
ave = np.average(y_train)
clf = MultinomialNB(class_prior=[1-ave, ave])とすると何故かいくらか分類され、1000試行の平均は次のようになったが、AUCが低いし、テストデータに対する性能値はWekaのBayesNetよりも常に低かった。clf = MultinomialNB(class_prior=[0.5, 0.5])
Average with training data Class FamilyOut=False: Precision=0.930, Recall=0.950, F-measure=0.940 Class FamilyOut=True : Precision=0.681, Recall=0.597, F-measure=0.635 Average with test data Class FamilyOut=False: Precision=0.930, Recall=0.950, F-measure=0.939 Class FamilyOut=True : Precision=0.676, Recall=0.590, F-measure=0.622 Average of AUC with training data=0.653 Average of AUC with test data=0.648
BernoulliNBとMultinomialNBはアルゴリズムが異なるが、これほどまでに性能が悪い理由も、family-out=TRUEの事前確率を0.5としない限り全く分類されない理由もわからない。
MultinomialNBはナイーブベイズ分類器の代表的なアルゴリズムの実装であり、これほど使いにくいものとは思えない。何か使い方がまずいのだろうか。
他のデータでも試したことがあるが、MultinomialNBはclass_priorを設定しない(自動計算にする)と大体妙な結果になるように思う。MultinomialNBはclass_priorの扱いが怪しいのだろうか。
なお、.arff形式のデータファイルは、次のコードで出力した。
# Save data in .arff format
import os
def save_arff(filename, data):
f = open(os.getenv("HOME") + "/tmp/" + filename, 'w')
f.write("""@relation familyout
@attribute family-out {0, 1}
@attribute bowel-problem {0, 1}
@attribute light-on {0, 1}
@attribute dog-out {0, 1}
@attribute hear-bark {0, 1}
@data
""")
for x in data:
f.write("{},{},{},{},{}\n".format(*x))
f.close()
save_arff("family_out_train.arff", train_data)
save_arff("family_out_test.arff", test_data)

コメント