前記事の続きである。
苦労の末に前記事のCompileErrorの問題をクリアして、「空間自己回帰モデル」に進んだら、そのすぐ後の状態空間モデルとCARモデルの比較でまたPyMCのトラブルに遭遇した。
まず、トラブルが起こらなかった方の、p.51〜p.52のIntrinsic CAR(Conditional Autoregressive Prior)モデルのサンプルコードをPyMCで書き直したものを貼る。
●データ
import pandas as pd
import matplotlib.pyplot as plt
# サンプルデータ
# https://kuboweb.github.io/-kubo/stat/iwanamibook/fig/spatial/Y.RData の中のYのみ
Y = [ 0, 3, 2, 5, 6, 16, 8, 14, 11, 10, 17, 19, 14, 19, 19, 18, 15,
13, 13, 9, 11, 15, 18, 12, 11, 17, 14, 16, 15, 9, 6, 15, 10, 11,
14, 7, 14, 14, 13, 17, 8, 7, 10, 4, 5, 5, 7, 4, 3, 1]
N = len(Y)
plt.scatter(range(1, N+1), Y)
plt.ylim(-1, 25)
plt.xlabel('position')
plt.ylabel('count')
plt.show()
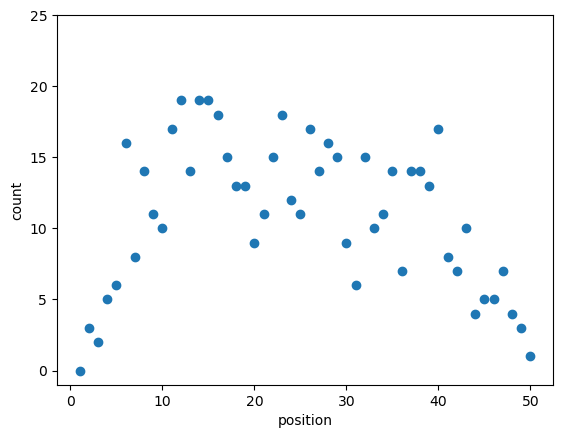
これは隣接する50地点の植物の個体数をカウントしたという想定で作られたデータとのことである。この観測値はポアソン分布に従うとし、各地点の平均にIntrinsic CARモデルを当てはめてみる。 幸いPyMCにもpymc.ICARというIntrinsic CARのライブラリがあったので、これを使った。
●コード
import numpy as np
import pymc as pm
# adjacency matrix(隣接行列)
adj = np.array([[j == i+1 or j == i-1 for i in range(N)] for j in range(N)]).astype(int)
with pm.Model() as model:
# beta = pm.Normal('beta', 0, 1e-4) # 本の通りだがこれだと全く平滑化されない、それっぽい結果の時のbetaは2より大きい
beta = pm.Normal('beta', 0, 100)
sigma = pm.Uniform('sigma', 0, 100)
# centered parameterization
# これだと全く平滑化されない
# S = pm.ICAR('S', W=adj, sigma=sigma)
# l = pm.Deterministic('lambda', (beta + S).exp())
# non-centered parameterization
S = pm.ICAR('S', W=adj)
l = pm.Deterministic('lambda', (beta + sigma * S).exp())
Yobs = pm.Poisson('Yobs', mu=l, observed=Y)
with model:
trace = pm.sample(1000, progressbar=False)
# 結果の可視化
import arviz as az
x = range(1, N+1)
plt.scatter(x, Y, label='observed')
plt.ylim(-1, 25)
lambda_mean_CAR = pm.summary(trace, var_names='lambda')['mean']
plt.plot(x, lambda_mean_CAR, color='r', linewidth=2, label='posterior lambda')
# 95%ベイズ信用区間
hdi_CAR = az.hdi(trace, hdi_prob=0.95, var_names='lambda')
plt.fill_between(x, hdi_CAR['lambda'][:,0], hdi_CAR['lambda'][:,1], color='pink', alpha=0.15, label='95% credible interval')
plt.xlabel('position')
plt.ylabel('count')
plt.legend()
plt.show()
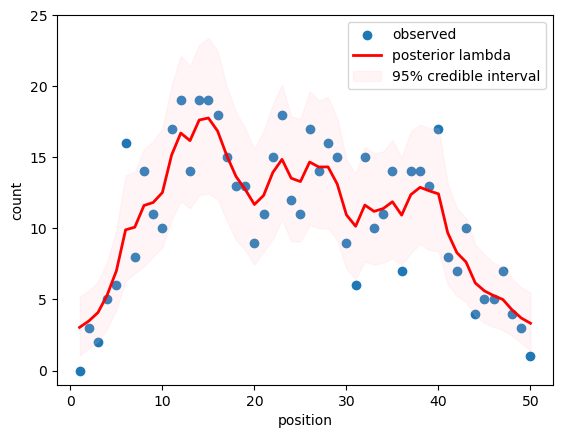
理屈がわかってない為に、平滑化されるまでに苦労したが、本と大体同じ結果が得られた。
次に、問題のp.55辺りの状態空間モデルの例である。1次元1階差分のIntrinsic CARモデルはローカルレベルモデルと等価とのことで、大体同じ結果になることを確認する。
●コード
%%time
# 発見個体数からのパラメーター推定
import pymc as pm
import pandas as pd
# 不具合回避用
# N = 31 # N > 31だとAttributeError: 'Scratchpad' object has no attribute 'ufunc'
with pm.Model() as model:
# 事前分布
alpha = [pm.Uniform('alpha[1]', 0, 10)]
sigma = pm.Uniform('sigma', 0, 100)
# システムモデル
for i in range(1, N):
alpha.append(pm.Normal(f'alpha[{i+1}]', mu=alpha[i-1], sigma=sigma))
# 観測モデル
for i in range(N):
lambda_tmp = pm.Deterministic(f'lambda[{i+1}]', alpha[i].exp())
Yobs = pm.Poisson(f'Y[{i+1}]', mu=lambda_tmp, observed=Y[i])
with model:
trace = pm.sample(1000, progressbar=False)
File ~/Library/Python/3.10/lib/python/site-packages/pytensor/tensor/elemwise.py:747, in Elemwise.perform(self, node, inputs, output_storage)
745 ufunc = self.ufunc
746 else:
--> 747 ufunc = node.tag.ufunc
748 else:
749 ufunc = node.tag.ufunc
File ~/Library/Python/3.10/lib/python/site-packages/pytensor/graph/utils.py:286, in Scratchpad.__getattribute__(self, name)
285 def __getattribute__(self, name):
--> 286 return super().__getattribute__(name)
AttributeError: 'Scratchpad' object has no attribute 'ufunc'
HINT: Re-running with most PyTensor optimizations disabled could provide a back-trace showing when this node was created. This can be done by setting the PyTensor flag 'optimizer=fast_compile'. If that does not work, PyTensor optimizations can be disabled with 'optimizer=None'.
HINT: Use the PyTensor flag `exception_verbosity=high` for a debug print-out and storage map footprint of this Apply node.
これまた、前回と同じく、N<=31ではエラーが発生しなかった。しかし、前回のようにg++のCompileErrorではないので、当然同じ対策は無効だった。
今度はエラーメッセージにも回避策に繋がるヒントがほとんど無い。Webで検索しても、同じエラーが出たという情報はあるが解決方法が全く見つからなかった。
最初に発生を確認したバージョンは Python 3.10.14 + PyMC 5.15.0 だが、PyMCのドキュメントに従ってMiniforgeのcondaを用いて Python 3.12.3 + PyMC 5.15.1 をインストールしても結果は同じだった。
発生箇所は、PyTensorのelemwise.pyのperform()のこの部分の最後の行である。
if self.ufunc:
ufunc = self.ufunc
elif not hasattr(node.tag, "ufunc"):
# It happen that make_thunk isn't called, like in
# get_underlying_scalar_constant_value
self.prepare_node(node, None, None, "py")
# prepare_node will add ufunc to self or the tag
# depending if we can reuse it or not. So we need to
# test both again.
if self.ufunc:
ufunc = self.ufunc
else:
ufunc = node.tag.ufunc # <--
この部分で、self.ufuncもnode.tag.ufuncも無ければself.prepare_node()を呼び出してself.ufuncかnode.tag.ufuncかを得ることになっているが、prepare_node()のコードでは、ある条件ではどちらにもufuncが入らないようになっている。
def prepare_node(self, node, storage_map, compute_map, impl):
# Postpone the ufunc building to the last minutes due to:
# - NumPy ufunc support only up to 32 operands (inputs and outputs)
# But our c code support more.
# - nfunc is reused for scipy and scipy is optional
if (len(node.inputs) + len(node.outputs)) > 32 and impl == "py":
impl = "c"
NumPyの制限により、引数の数が32を超える場合はNumPyでなく"c code"を使うと書かれているが、impl = "c"の場合、この後selfにもnode.tagにもufuncが入らない。
perform()の最初の方のコメントにも
def perform(self, node, inputs, output_storage):
if (len(node.inputs) + len(node.outputs)) > 32:
# Some versions of NumPy will segfault, other will raise a
# ValueError, if the number of operands in an ufunc is more than 32.
# In that case, the C version should be used, or Elemwise fusion
# should be disabled.
# FIXME: This no longer calls the C implementation!
super().perform(node, inputs, output_storage)
最新のPyMCのコードを確認しても、この辺りのコードに変化は無かった。
super().perform() は OpenMPOp.perform() のようなので、OpenMPを有効にすれば変わるかも知れないと思ったが、 ~/.pytensorrc で openmp = True にしてもWarningが出てFalseになった。
"Elemwise fusion"を停止するには、この辺りを見ると ~/.pytensorrc で optimizer = None とか optimizer_excluding = elemwise_fusion とかとすると良さそうだが、結果は同じだった。
前回と同様、システムモデルの部分にpm.GaussianRandomWalkを使えば何の問題も起こらなかったが、それでは本のサンプルコードと形が違い過ぎるし、状態空間モデルをPyMCで直接的に書きたくてこの問題に遭遇した場合に解決しないので、満足できなかった。
●コード
# 発見個体数からのパラメーター推定(pm.GaussianRandomWalk使用)
import pymc as pm
import pandas as pd
N = 50
with pm.Model() as model:
# 事前分布
sigma = pm.Uniform('sigma', 0, 100)
# システムモデル
alpha = pm.GaussianRandomWalk('alpha', mu=0, sigma=sigma, init_dist=pm.Uniform.dist(0, 10), steps=N-1)
# 観測モデル
l = pm.Deterministic(f'lambda', alpha.exp())
for i in range(N):
Yobs = pm.Poisson(f'Y[{i+1}]', mu=l[i], observed=Y[i])
initvals = {}
with model:
trace = pm.sample(1000, initvals=initvals, progressbar=False)
# 結果の可視化
import arviz as az
import matplotlib.pyplot as plt
x = range(1, N+1)
plt.scatter(x, Y, label='observed')
plt.ylim(-1, 25)
# ICARを用いた結果(比較用)
plt.plot(x, lambda_mean_CAR, color='r', linewidth=2, label='posterior lambda')
plt.fill_between(x, hdi_CAR['lambda'][:,0], hdi_CAR['lambda'][:,1], color='pink', alpha=0.15, label='95% credible interval')
# 状態遷移モデルを用いた結果
lambda_mean_RW = pm.summary(trace, var_names='lambda')['mean']
plt.plot(x, lambda_mean_RW, color='b', linewidth=2, label='posterior lambda')
# 95%ベイズ信用区間
hdi_RW = az.hdi(trace, hdi_prob=0.95, var_names='lambda')
plt.fill_between(x, hdi_RW['lambda'][:,0], hdi_RW['lambda'][:,1], color='cyan', alpha=0.15, label='95% credible interval')
plt.xlabel('position')
plt.ylabel('count')
plt.legend()
plt.show()
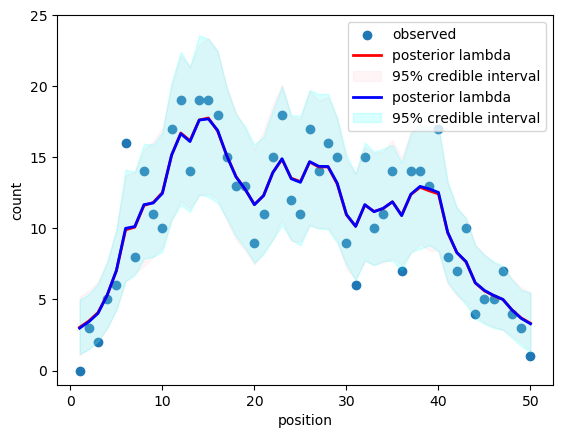
一応、Intrinsic CARの結果と状態空間モデルの結果がほぼ一致することが確認できた。
これでは納得できなかったので、PyMC Discourseというコミュニティで質問をしてみたら、pytensor.scanを使えば良い(そもそもPyMCのモデルではループを使うべきでない)という回答を得た。
AttributeError: 'Scratchpad' object has no attribute 'ufunc' - Questions / v5 - PyMC Discourse
この投稿者のmaruinenは筆者で、やんわりとPyTensorの不具合について指摘したつもりだったが、それについては誰からもコメントされなかった。
このやり取りにあるように、何とかpytensor.scanを用いた状態空間モデルの書き方を理解して、上のAttributeErrorが出た例を書き直して動かすことに成功した。
●コード
# 発見個体数からのパラメーター推定(pytensor.scanを使う例)
import pymc as pm
import pandas as pd
from pymc.pytensorf import collect_default_updates
import pytensor
import pytensor.tensor as pt
N = 50
# Helper function for pm.CustomDist
n_steps = N - 1
def statespace_dist(mu_init, sigma_level, size):
def grw_step(mu_tm1, sigma_level):
mu_t = mu_tm1 + pm.Normal.dist(sigma=sigma_level)
return mu_t, collect_default_updates(outputs=[mu_t])
mu, updates = pytensor.scan(fn=grw_step,
outputs_info=[{"initial": mu_init}],
non_sequences=[sigma_level],
n_steps=N-1,
name='statespace',
strict=True)
return mu
with pm.Model() as model:
# 事前分布
alpha1 = pm.Uniform('alpha[1]', 0, 10)
sigma = pm.Uniform('sigma', 0, 100)
# システムモデル
alpha = pm.CustomDist('alpha', alpha1, sigma, dist=statespace_dist)
alpha = pt.concatenate([[alpha1], alpha])
# 観測モデル
l = pm.Deterministic('lambda', alpha.exp())
Yobs = pm.Poisson('Y', mu=l, observed=Y)
with model:
trace = pm.sample(1000, progressbar=False)
# 結果の可視化
import arviz as az
import matplotlib.pyplot as plt
x = range(1, N+1)
plt.scatter(x, Y, label='observed')
plt.ylim(-1, 25)
# ICARを用いた結果(比較用)
plt.plot(x, lambda_mean_CAR, color='r', linewidth=2, label='posterior lambda')
plt.fill_between(x, hdi_CAR['lambda'][:,0], hdi_CAR['lambda'][:,1], color='pink', alpha=0.15, label='95% credible interval')
# 状態遷移モデルを用いた結果
lambda_mean_SS = pm.summary(trace, var_names='lambda')['mean']
plt.plot(x, lambda_mean_SS, color='b', linewidth=2, label='posterior lambda')
# 95%ベイズ信用区間
hdi_SS = az.hdi(trace, hdi_prob=0.95, var_names='lambda')
plt.fill_between(x, hdi_SS['lambda'][:,0], hdi_SS['lambda'][:,1], color='cyan', alpha=0.15, label='95% credible interval')
plt.xlabel('position')
plt.ylabel('count')
plt.legend()
plt.show()
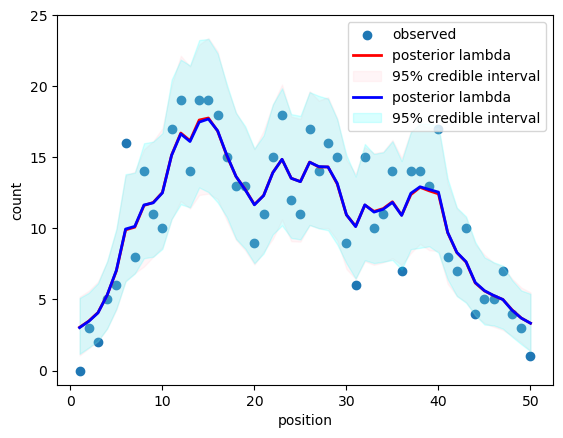
PyMCを使い始めていきなり、こんなトラブルで調べまくる羽目になったのは辛かった。それに、ループを使うべきではない、代わりにpytensor.scanを使うべきだとされているが、PyMCでpytensor.scanを使うのは難し過ぎる。PyMCは結構歴史が古いようだが、まだまだ完成度が低いのだろうか。
上に書いたエラー以外にも、SamplingErrorなど、MCMC法のサンプリングが発散してエラーになったり、極端に大きな値になるなど、正常な結果が得られないことも多発した。初期値や事前確率のパラメーターを調整すると正常な結果が得られたこともあったが、pt.config.optimizerを変更するとエラーになったりならなかったりすることもあった。
一度、.pytensorrcに"optimizer = None"と書いたまま消し忘れて、動いた実績のあるコードが動かなくなって、原因の調査に数時間を取られたことがあった。
そのようなことはPyMCに限らずよくあることで、機械学習系のライブラリを使うにはトラブルに悩まされて想像以上の時間を奪われる覚悟が必要だと、改めて思った。
コメント