import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
# サンプルデータ作成
np.random.seed(666)
df = pd.DataFrame({'value': np.random.randn(365).cumsum()},
index=pd.date_range('2019-1-1', periods=365))
df
| value | |
|---|---|
| 2019-01-01 | 0.824188 |
| 2019-01-02 | 1.304154 |
| 2019-01-03 | 2.477622 |
| 2019-01-04 | 3.386670 |
| 2019-01-05 | 2.814949 |
| ... | ... |
| 2019-12-27 | -1.935362 |
| 2019-12-28 | -1.170606 |
| 2019-12-29 | -0.112895 |
| 2019-12-30 | 0.490068 |
| 2019-12-31 | -0.184056 |
# 描画
fig = plt.figure(figsize=(15, 3))
df['value'].plot()
plt.axhline(0, color='r')
plt.show()
こういうデータがあり、値が0より大きい日が5日以上連続する区間をグラフ上で示したいとする。
筆者は過去に似たようなことをしたい時があり、適当な方法がわからなかったので、効率が悪いと知りつつ、次のように、DataFrameの各行をループ処理で1行ずつ調べて該当区間を求めるようにした。
# 'value' > 0 が5日以上連続する区間を求める
df['cont_days'] = 0 # 'value' > 0 が連続する日数
df['ge_5d'] = False # 連続する日数が5日以上(greater than or equal to)かどうか
flag = False # 1つ前が 'value' > 0 がどうか
for i in range(len(df)):
if df.iloc[i]['value'] > 0:
if flag == False:
start_i = i # value' > 0 の開始位置を保存
flag = True
else:
if flag == True:
end_i = i # value' > 0 の終了位置
if end_i - start_i >= 5:
print("{} - {} ({} days)".format(
df.index[start_i].date(), df.index[end_i - 1].date(), end_i - start_i))
df.loc[df.index[start_i:end_i], ['cont_days', 'ge_5d']] = end_i - start_i, (end_i - start_i >= 5)
flag = False
df
2019-01-01 - 2019-01-16 (16 days) 2019-03-17 - 2019-03-29 (13 days) 2019-04-20 - 2019-05-01 (12 days) 2019-05-14 - 2019-05-26 (13 days) 2019-06-12 - 2019-07-20 (39 days) 2019-07-22 - 2019-07-26 (5 days) 2019-07-29 - 2019-08-04 (7 days) 2019-08-13 - 2019-10-18 (67 days)
| value | cont_days | ge_5d | |
|---|---|---|---|
| 2019-01-01 | 0.824188 | 16 | True |
| 2019-01-02 | 1.304154 | 16 | True |
| 2019-01-03 | 2.477622 | 16 | True |
| 2019-01-04 | 3.386670 | 16 | True |
| 2019-01-05 | 2.814949 | 16 | True |
| ... | ... | ... | ... |
| 2019-12-27 | -1.935362 | 0 | False |
| 2019-12-28 | -1.170606 | 0 | False |
| 2019-12-29 | -0.112895 | 0 | False |
| 2019-12-30 | 0.490068 | 0 | False |
| 2019-12-31 | -0.184056 | 0 | False |
# 描画
fig = plt.figure(figsize=(15,3))
ax1 = fig.gca()
# 'value' > 0 が5日以上連続する区間を塗り潰す
ax2 = ax1.twinx()
ax2.fill_between(df.index, 0, df['ge_5d'], color='r', alpha=0.2, linewidth=0, step='post')
ax2.axes.yaxis.set_visible(False)
# 'value'の描画、先にするとX軸のラベルのフォーマットが変わるので後でする
df['value'].plot(ax=ax1)
ax1.axhline(color='r')
plt.show()
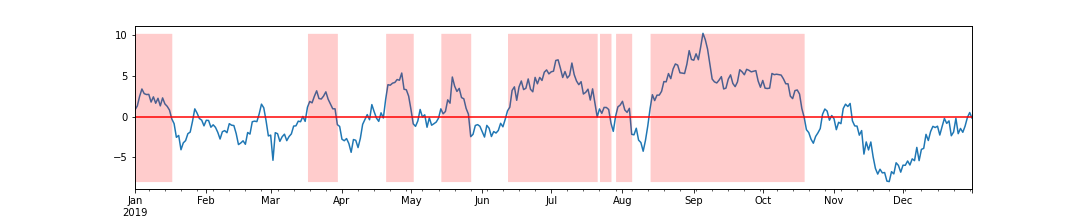
後日、そういうのは次のようにshift()とcumsum()をうまく使えばgroupby()で処理できるということを教えてもらった。
●改良後のコード
# 'value' > 0 が5日以上連続する区間を求める
df['flag'] = df['value'] > 0
df['cont_days'] = df.groupby((df['flag'] != df['flag'].shift()).cumsum())['flag'].transform(sum)
df['ge_5d'] = df['cont_days'] >= 5
df
| value | flag | cont_days | ge_5d | |
|---|---|---|---|---|
| 2019-01-01 | 0.824188 | True | 16 | True |
| 2019-01-02 | 1.304154 | True | 16 | True |
| 2019-01-03 | 2.477622 | True | 16 | True |
| 2019-01-04 | 3.386670 | True | 16 | True |
| 2019-01-05 | 2.814949 | True | 16 | True |
| ... | ... | ... | ... | ... |
| 2019-12-27 | -1.935362 | False | 0 | False |
| 2019-12-28 | -1.170606 | False | 0 | False |
| 2019-12-29 | -0.112895 | False | 0 | False |
| 2019-12-30 | 0.490068 | True | 1 | False |
| 2019-12-31 | -0.184056 | False | 0 | False |
改良後のコード中の
groupby((df['flag'] != df['flag'].shift()).cumsum())
は初見ではややこしいが、次の例で説明すると、df['flag'].shift()が1つ前の値、df['flag'] != df['flag'].shift()が1つ前と同じかどうかで、それを累積(cumsum)することにより、'flag'が前と同じ値の所は同じ番号、変化があった所で次の番号となり、これをgroupby()のキーにすることにより、'flag'の同じ値が連続する区間毎にグループ分けされる。
# groupby((df['flag'] != df['flag'].shift()).cumsum()) の解説用
df = pd.DataFrame({
'flag': [False, False, True, True, False, True, True, True, False, False]})
df['shift'] = df['flag'].shift()
df['diff'] = df['flag'] != df['shift']
df['cont_group'] = df['diff'].cumsum()
df
| flag | shift | diff | cont_group | |
|---|---|---|---|---|
| 0 | False | NaN | True | 1 |
| 1 | False | False | False | 1 |
| 2 | True | False | True | 2 |
| 3 | True | True | False | 2 |
| 4 | False | True | True | 3 |
| 5 | True | False | True | 4 |
| 6 | True | True | False | 4 |
| 7 | True | True | False | 4 |
| 8 | False | True | True | 5 |
| 9 | False | False | False | 5 |
改良前のコードと改良後のコードを比較すると、改良後のコードは断然短いし、処理時間も圧倒的に短く(筆者の環境では改良前約200ms、改良後約7.5ms)、しかもデータサイズが100倍になっても処理時間が少ししか伸びない(改良前約14秒、改良後約12.5ms)。
教えてもらった所の他の人のコメントを見ると、その筋では「shiftを使えばいい」だけで以上のことが通じるらしいことになっていた。
pandas documentationの"Cookbook"の"Grouping like Python's itertools.groupby"の所に載っているし、stackoverflowのあるページに"uses some common idioms"と書かれているので、きっとよく知られたパターンなのだろう。
なお、改良後のコード中の (df['flag'] != df['flag'].shift()) の部分は、 df['flag'].diff() としても同じ結果になりそうだが、後者は先頭行がNaNになるので、同じ結果にならない。
なお、改良前のコードでは、値が0より大きい日が5日以上連続する区間の最初の日と最後の日を表示しているが、改良後のコードの方法で同様のことをやってみたものを一例として以下に貼り付ける。
# 'value' > 0 が5日以上連続する区間の初日と最終日を表示する
df['flag'] = df['value'] > 0
tmpdf = df.groupby((df['flag'] != df['flag'].shift()).cumsum())['flag'].agg(
start = lambda x: x.index[0].date(),
end = lambda x: x.index[-1].date(),
days = sum)
tmpdf[tmpdf['days'] >= 5].style.hide_index()
| start | end | days |
|---|---|---|
| 2019-01-01 | 2019-01-16 | 16 |
| 2019-03-17 | 2019-03-29 | 13 |
| 2019-04-20 | 2019-05-01 | 12 |
| 2019-05-14 | 2019-05-26 | 13 |
| 2019-06-12 | 2019-07-20 | 39 |
| 2019-07-22 | 2019-07-26 | 5 |
| 2019-07-29 | 2019-08-04 | 7 |
| 2019-08-13 | 2019-10-18 | 67 |
コメント